“Io tengo il soggetto della mia indagine costantemente davanti a me ed aspetto fino a quando i primi albori si sviluppano gradualmente poco a poco fino a diventare una luce chiara e piena.”
— Sir Isaac Newton
Le cellule eucariotiche possiedono una straordinaria capacità di coordinare processi complessi grazie ai contatti tra membrane (MCSs), punti di interazione tra organelli cellulari che facilitano lo scambio di molecole e segnali. Comprendere la dinamica di questi contatti è cruciale per svelare i meccanismi alla base di malattie come il cancro e le neurodegenerazioni. Tuttavia, i limiti delle tecniche attuali hanno reso difficile ottenere un quadro completo. In questo contesto, un team di ricercatori ha sviluppato una nuova generazione di sonde chimogenetiche per osservare e misurare in simultanea la morfologia e la funzionalità degli MCSs.
García Casas, P., Rossini, M., Påvénius, L. et al. Simultaneous detection of membrane contact dynamics and associated Ca2+ signals by reversible chemogenetic reporters. Nat Commun 15, 9775 (2024). https://doi.org/10.1038/s41467-024-52985-0
L’importanza dei contatti tra membrane nelle cellule
Gli MCSs rappresentano piattaforme specializzate dove avvengono processi cellulari fondamentali come il trasferimento di calcio (Ca²⁺) e il metabolismo lipidico. La loro prossimità permette uno scambio efficiente di informazioni e metaboliti tra organelli come il reticolo endoplasmatico (ER) e i mitocondri. Disfunzioni in queste interazioni sono state associate a malattie neurodegenerative e tumori, sottolineando l’importanza di approfondire la loro conoscenza. Tuttavia, le dimensioni nanometriche e la natura dinamica degli MCSs hanno reso questa impresa una sfida per la scienza.
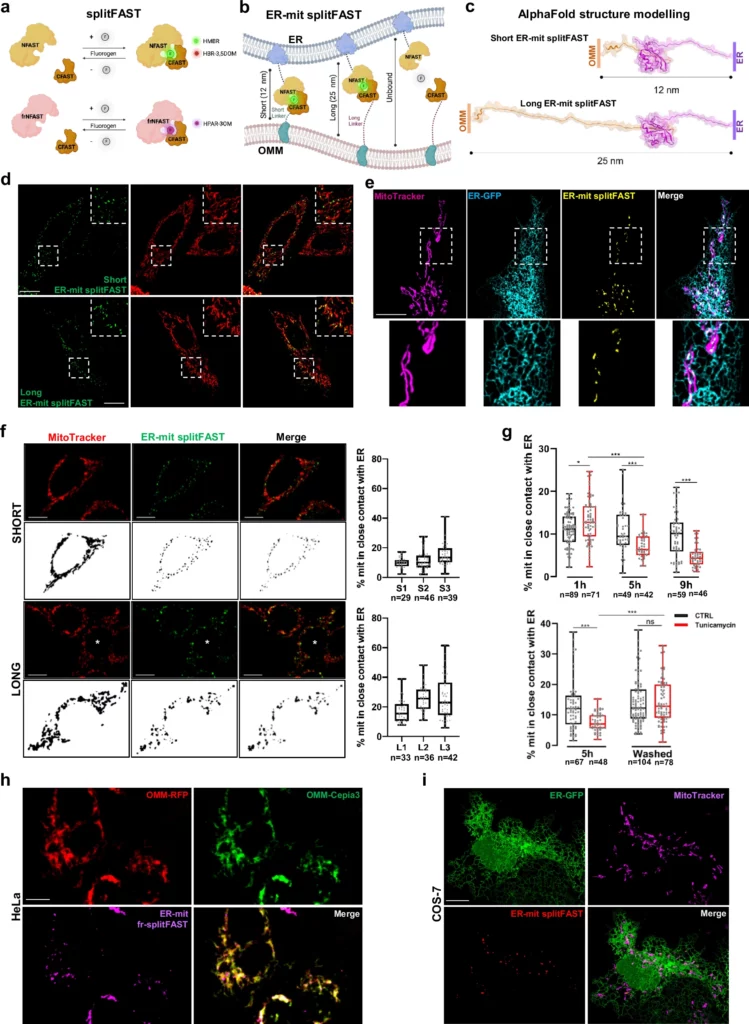
a La vignetta rappresenta il sistema splitFAST, formato dalle porzioni NFAST e CFAST, che non sono di per sé fluorescenti, a meno che non siano assemblate e legate a un fluorogeno: verde, rosso o rosso lontano (quest'ultimo richiede frNFAST). Creato in BioRender. b, c La vignetta (b) rappresenta l'adattamento di splitFAST per marcare gli MCS ER-mit. Creato in BioRender. Indirizzando la porzione NFAST alla membrana ER e CFAST10 all'OMM, entrambe separate da linker non strutturati di dimensioni diverse come modellato da AlphaFold (c), sono state generate due diverse sonde per studiare le MCS ER-mit: la splitFAST corta ER-mit, con una lunghezza di ~12 nm, e la splitFAST lunga ER-mit, di ~25 nm. Le strutture AlphaFold proposte forniscono modelli statici delle sonde in una conformazione estesa, ma i linker non strutturati possono piegarsi e adottare conformazioni multiple (vedi Metodi). d Immagini confocali rappresentative di cellule HeLa che coesprimono la ER-mit splitFAST corta o lunga (che mostra un pattern punteggiato lungo i mitocondri) con una mit-RFP. e Immagine confocale rappresentativa di una cellula COS-7 che coesprime l'ER-mit splitFAST corto e una ER-GFP, colorata con MitoTracker Deep Red. Un segnale punteggiato di ER-mit splitFAST colocalizza sia con l'ER che con i mitocondri, marcando i siti di stretto contatto tra i due organelli. f Immagini confocali rappresentative di cloni stabili di cellule HeLa che esprimono la versione corta (S1, S2, S3) o lunga (L1, L2, L3) delle sonde ER-mit splitFAST. È mostrato un segnale tratteggiato lungo i mitocondri (etichettati con MitoTracker Deep Red), che consente di calcolare la percentuale della superficie mitocondriale (% mit) coperta da contatti corti o lunghi con l'ER (box plot a destra, vedi “Metodi”). La cellula indicata con un asterisco bianco è mostrata nella corrispondente immagine binaria utilizzata per l'analisi. g I box plot rappresentano i cambiamenti dinamici dei MCS ER-mit (calcolati come nel pannello (f)) in cellule HeLa che esprimono lo splitFAST ER-mit corto, dopo trattamenti con tunicamicina (5 μg/ml). Il recupero dei livelli basali di MCS ER-mit è stato osservato dopo la rimozione della tunicamicina. h, i Immagini confocali rappresentative di (h) cellule HeLa che coesprimono OMM-RFP, OMM-Cepia3 e la sonda ER-mit corta fr-splitFAST, o (i) di cellule COS-7 che coesprimono ER-GFP, ER-mit corta splitFAST e in cui i mitocondri sono stati marcati con MitoTracker Deep Red. Barra di scala: 10 μm (d-f, h, i).
Limitazioni delle tecniche attuali
Le tecniche di imaging come la microscopia ottica e quella elettronica offrono vantaggi distinti ma presentano limiti significativi. La microscopia ottica, pur permettendo di seguire i cambiamenti nel tempo, non può risolvere dettagli nanometrici. La microscopia elettronica, invece, offre risoluzione spaziale elevata ma è limitata ai campioni fissi. Metodi più recenti, come le sonde fluorescenti basate su FRET, offrono nuove opportunità, ma spesso richiedono attrezzature complesse e non riescono a catturare la dinamicità degli MCSs in tempo reale.
Una soluzione innovativa: le sonde chimogenetiche reversibili
Il team di ricerca ha sviluppato sonde chimogenetiche basate su splitFAST, un sistema che utilizza frammenti proteici per generare segnali fluorescenti solo quando vicini. Questa tecnologia permette di osservare in tempo reale la formazione e il dissolvimento degli MCSs senza interferire con la loro dinamica naturale. Inoltre, l’integrazione di moduli sensibili al calcio (come PRINCESS) consente di monitorare contemporaneamente i segnali di Ca²⁺, offrendo una visione integrata della morfologia e della funzionalità degli MCSs.
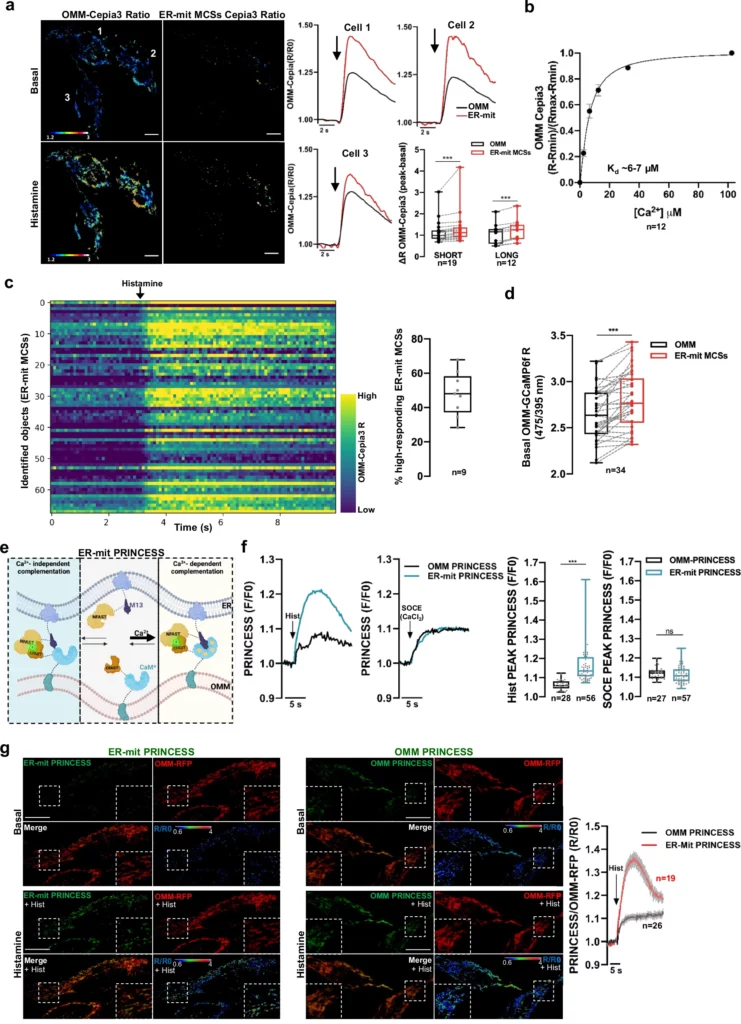
a Immagini rappresentative del rapporto 475/390 nm (R, vedi Metodi) di OMM-Cepia3, co-espresso in cellule HeLa con ER-mit RspA-splitFAST corto o lungo per marcare MCS ER-mit corto o lungo. Sono mostrate le immagini del rapporto tra OMM-Cepia3 e la porzione di OMM-Cepia3 co-localizzata con ER-mit RspA-splitFAST (ER-mit MCSs Cepia3 Ratio), prima (basale) e dopo la stimolazione con istamina (100 µM). A destra, i tracciati rappresentano i rapporti di OMM-Cepia3 (OMM) o della porzione di OMM-Cepia3 co-localizzata con ER-mit RspA-splitFAST corto (ER-mit) alla stimolazione con istamina (frecce), per le tre cellule a sinistra. I box plots rappresentano il ΔR di OMM-Cepia3 alla stimolazione con istamina sia nel bulk OMM che nelle regioni co-localizzate con MCS ER-mit, in cellule che esprimono RspA-splitFAST corto o lungo ER-mit, come indicato.
b OMM-Cepia3, espresso in cellule HeLa, è stato calibrato permeabilizzando le cellule con digitonina (25 µM) in un tampone intracellulare e aggiungendo concentrazioni fisse di Ca2+. Sono state eseguite misurazioni raziometriche a 475/390 nm (vedi Metodi). Media ± SEM. c La mappa di calore rappresenta il rapporto OMM-Cepia3 nel tempo nelle sottoregioni co-localizzate con i MCS ER-mit corti (identificati come oggetti singoli, asse y) di una cellula HeLa (come in a). Alla stimolazione con istamina, il 50% delle MCS ER-mit presenta elevate concentrazioni di Ca2+ (box plot a destra). d Il box plot rappresenta il rapporto 475/390 nm di OMM-GCaMP6f in cellule HeLa in condizioni basali, sia nell'OMM di massa sia in corrispondenza di MCS ER-mit corti (identificati dalla co-espressione e dalla co-localizzazione con ER-mit RspA-splitFAST). e La vignetta rappresenta il razionale alla base della progettazione di ER-mit PRINCESS. La Calmodulina mutata (CaM*) di Cepia3 e il peptide M13 sono stati incorporati in ER-mit splitFAST, per fornire capacità di rilevamento del Ca2+. Creato in BioRender.
f Tracciati rappresentativi della fluorescenza di ER-mit PRINCESS o OMM-PRINCESS in cellule HeLa, in seguito a stimolazione con istamina (100 µM) in mKRB privo di Ca2+, o all'aggiunta di CaCl2 (2 mM) (SOCE) dopo 6 minuti di esaurimento del contenuto di Ca2+ dell'ER (ottenuto mediante stimolazione con istamina e thapsigargina (100 nM) in mKRB privo di Ca2+). A destra, i box-plot rappresentano i picchi di fluorescenza di OMM- o ER-mit PRINCESS (espressi come F/F0) dopo i trattamenti indicati. g Immagini confocali rappresentative di cellule HeLa che coesprimono OMM-RFP (usato per normalizzare il segnale fluorescente) con ER-mit PRINCESS o OMM-PRINCESS, prima (al basale) o dopo la stimolazione con istamina (100 µM) in mKRB privo di Ca2+. Sono mostrate anche le immagini del rapporto PRINCESS/OMM-RFP. A destra, sono mostrati i corrispondenti tracciati (media ± SEM) dei segnali di rapporto per le condizioni indicate. Barra di scala: 10 µm (a, g).
Applicazioni in modelli di malattia
Le sonde splitFAST sono state utilizzate per analizzare alterazioni degli MCSs in modelli di Alzheimer, dimostrando un aumento dell’interazione tra ER e mitocondri in condizioni patologiche. Questo strumento ha rivelato che l’accumulo di placche amiloidi altera il trasferimento di Ca²⁺, evidenziando un possibile meccanismo alla base della malattia. In modelli in vitro e in vivo, le sonde hanno mostrato una sensibilità senza precedenti, aprendo la strada a nuove scoperte nel campo della biologia cellulare.
Prospettive future e impatti sulla ricerca biomedica
L’introduzione delle sonde PRINCESS rappresenta un punto di svolta per lo studio degli MCSs. Grazie alla loro versatilità, queste tecnologie potrebbero essere adattate per studiare una vasta gamma di malattie e processi cellulari. Inoltre, l’approccio combinato di imaging ad alta risoluzione e misurazione funzionale potrebbe accelerare lo sviluppo di terapie mirate, offrendo nuove opportunità per la medicina personalizzata.
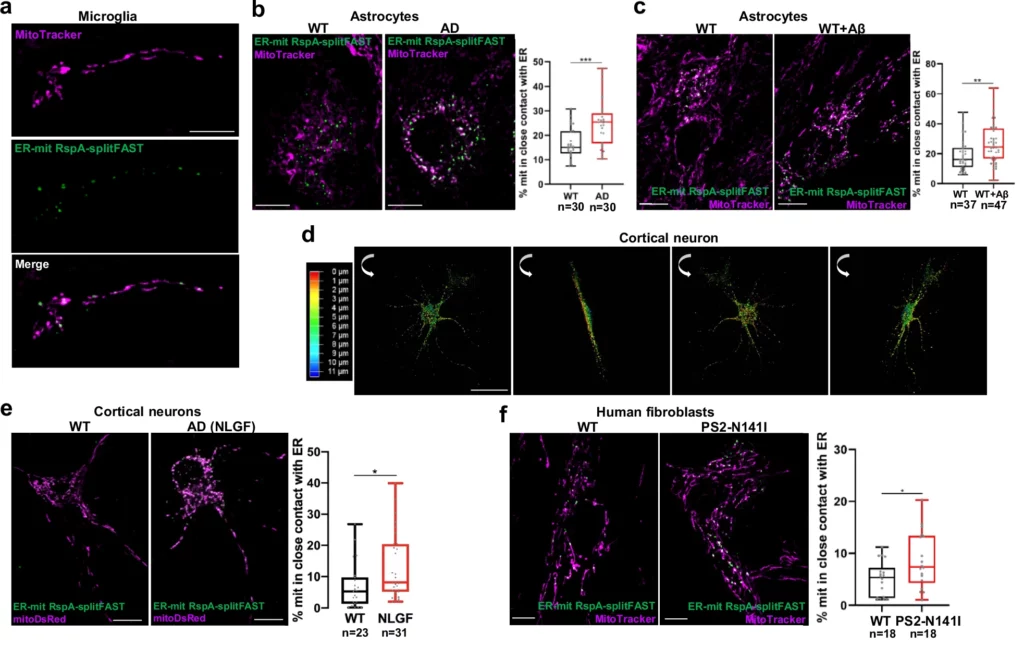
a-f Immagini confocali rappresentative di diverse cellule ER-mit RspA-splitFAST. Dove indicato, le cellule sono state colorate con MitoTracker Deep Red o mitoDsRed, utilizzato come marcatore mitocondriale. Le MCS ER-mit sono mostrate in microglia primaria di topo (a), astrociti (b, c), neuroni corticali (d, e) e fibroblasti umani (f). In (b, c, e, f) i box plot rappresentano la percentuale di superficie mitocondriale co-localizzata con ER-mit RspA-splitFAST per i tipi di cellule indicati. In b sono stati confrontati astrociti di topi WT o AD. In (c) gli astrociti di topi WT sono stati esposti (WT + Aβ) o meno (WT) a un mezzo condizionato contenente peptidi Aβ generati naturalmente. In (e) sono stati confrontati neuroni corticali di topi WT o AD (AppNL-G-F, abbreviato in NLGF).
In (f) sono stati confrontati fibroblasti cutanei umani primari provenienti da un donatore sano (WT) o da un paziente con AD familiare (PS2-N141I). *p < 0,05; **p < 0,01; ***p < 0,001. d Microscopia confocale proiezione 3D del segnale di fluorescenza ER-mit RspA-splitFAST in un neurone corticale di topi WT. La barra colorata a sinistra rappresenta la profondità (in µm) lungo l'asse z. Barra di scala: 10 µm (a-c, e, f); 50 µm (d).
In conclusione…
La scoperta di strumenti chimogenetici come splitFAST e PRINCESS rappresenta un passo avanti significativo nella comprensione delle interazioni cellulari. Questi avanzamenti non solo forniscono una comprensione più dettagliata della biologia degli MCSs, ma aprono anche nuove strade per la diagnosi e il trattamento di malattie complesse. Grazie a queste innovazioni, la scienza è più vicina a svelare i misteri delle reti cellulari che regolano la vita.





