“L’obesità è uno stato della psiche, una malattia causata dalla noia e dalla disillusione.”
— Alberto Lodispoto
L’obesità è una delle principali sfide sanitarie globali, correlata a malattie metaboliche, cardiovascolari e persino tumorali. Nonostante l’efficacia di interventi come la chirurgia bariatrica, il mantenimento della perdita di peso rimane una difficoltà per molti pazienti. Studi recenti rivelano che il tessuto adiposo può conservare una memoria epigenetica dell’obesità, ostacolando i benefici metabolici e promuovendo il recupero del peso. Questo articolo esplora come tali meccanismi epigenetici influenzano il tessuto adiposo e aprono nuove prospettive per trattamenti più efficaci.
Hinte, L.C., Castellano-Castillo, D., Ghosh, A. et al. Adipose tissue retains an epigenetic memory of obesity after weight loss. Nature (2024). https://doi.org/10.1038/s41586-024-08165-7
Obesità e memoria metabolica: una relazione complessa
L’obesità non è solo una condizione reversibile attraverso la perdita di peso. Anche dopo interventi significativi, come la chirurgia bariatrica, il tessuto adiposo subisce modifiche cellulari e trascrizionali che persistono. Queste alterazioni, definite memoria metabolica, suggeriscono che il corpo “ricorda” stati di obesità pregressi, influenzando negativamente il metabolismo futuro. Le scoperte mostrano che questa memoria non è confinata al tessuto adiposo, ma coinvolge anche altri compartimenti, come il sistema immunitario e l’endotelio vascolare.
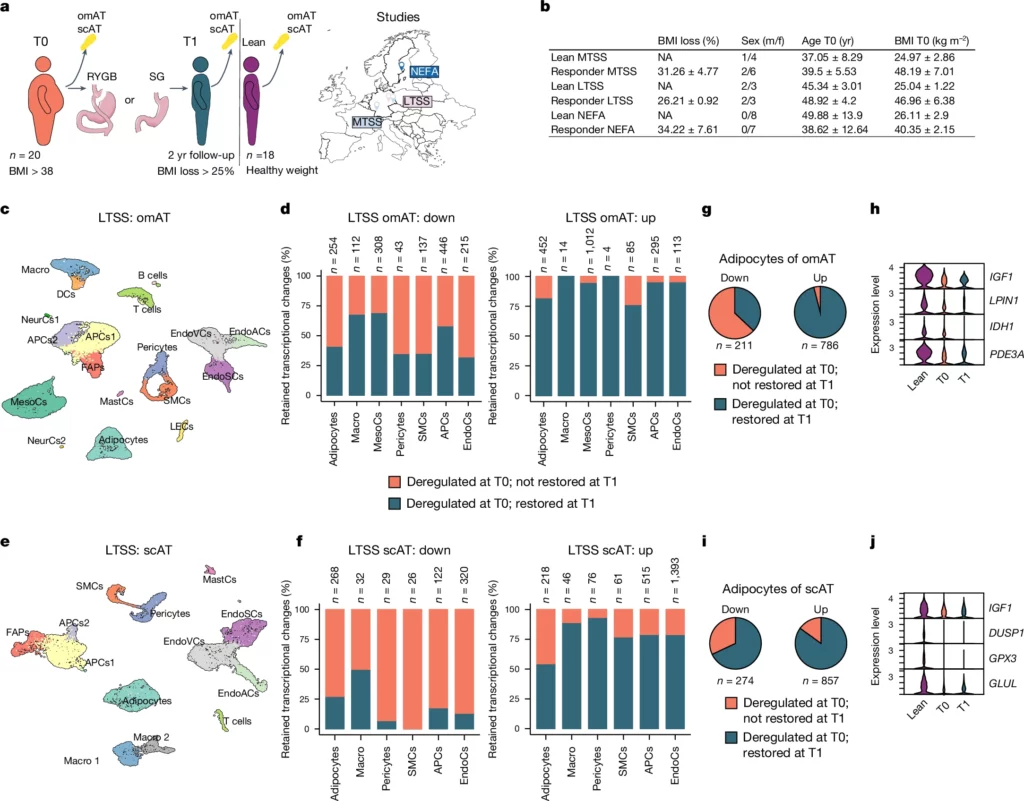
a, le biopsie di omAT e scAT sono state raccolte da persone affette da obesità durante il BaS (T0) e 2 anni dopo l'intervento (T1). Sono stati inclusi solo gli individui che avevano perso almeno il 25% del BMI rispetto a T0. Le biopsie di omAT e scAT sono state raccolte da individui sani e magri provenienti dagli stessi studi (MTSS, LTSS e NEFA). b, Sesso, età, BMI iniziale e perdita di BMI dei donatori magri e dei donatori con obesità. c, Uniform manifold approximation and projection (UMAP) di 22.742 nuclei che rappresentano pool di omAT di soggetti magri (n= 5; 2 maschi, 3 femmine) e omAT accoppiati di T0 e T1 (n= 5 ciascuno; 2 maschi, 3 femmine) di LTSS. d, Proporzione di cambiamenti trascrizionali conservati in tipi cellulari altamente abbondanti di omAT di LTSS. e, UMAP di 15.347 nuclei che rappresentano pool di scAT di soggetti magri (n= 5; 2 maschi, 3 femmine) e scAT accoppiati di T0 e T1 (n= 5 ciascuno; 2 maschi, 3 femmine) di LTSS. f, Proporzione di cambiamenti trascrizionali conservati in tipi di cellule altamente abbondanti di LTSS scAT. g, Proporzione di cambiamenti trascrizionali conservati negli adipociti omAT integrati di LTSS e MTSS omAT. h, Espressione normalizzata di DEG di memoria selezionati negli adipociti omAT. i, Proporzione di cambiamenti trascrizionali conservati negli adipociti omAT integrati di LTSS e NEFA scAT. j, espressione normalizzata dei DEG della memoria selezionati negli adipociti scAT. Per l'identificazione dei DEG in d, f, g, h, i e j è stato utilizzato il test Wilcoxon rank-sum con P< 0,01 aggiustato con il metodo di correzione di Bonferroni e log2 fold change (log2FC) > ±0,5 . DCs, cellule dendritiche; EndoCs, cellule endoteliali; EndoACs, EndoCs arteriolari; EndoSCs, EndoCs peduncolari; EndoVCs, EndoCs venulari; LECs, EndoCs linfatiche; FAPs, progenitori fibro-adipogenici; Macro, macrofagi; MastCs, mastociti; MesoCs, cellule mesoteliali; NeurCs, cellule simil-neuronali; SMCs, cellule muscolari lisce (vascolari); NA, non applicabile; m/f, maschio/femmina.
Cambiamenti epigenetici nel tessuto adiposo
Attraverso tecniche avanzate di sequenziamento, i ricercatori hanno identificato alterazioni epigenetiche stabili nei geni del tessuto adiposo. In particolare, il DNA degli adipociti presenta modifiche a livello delle istone, responsabili della regolazione genica. Tali modifiche persistono anche dopo una significativa perdita di peso e contribuiscono a mantenere un profilo trascrizionale pro-obesogeno, influenzando negativamente la funzione adipocitaria e promuovendo fenomeni infiammatori.
La plasticità del tessuto adiposo e il ruolo degli adipociti
Gli adipociti non sono semplicemente cellule di stoccaggio lipidico; svolgono ruoli chiave nella regolazione metabolica. Studi su modelli animali hanno dimostrato che gli adipociti mantengono modifiche epigenetiche anche dopo un cambio di dieta. Questa plasticità ha un lato oscuro: predispone il tessuto adiposo a rispondere in modo anomalo a stimoli nutrizionali successivi, favorendo il recupero del peso e lo sviluppo di complicanze metaboliche.
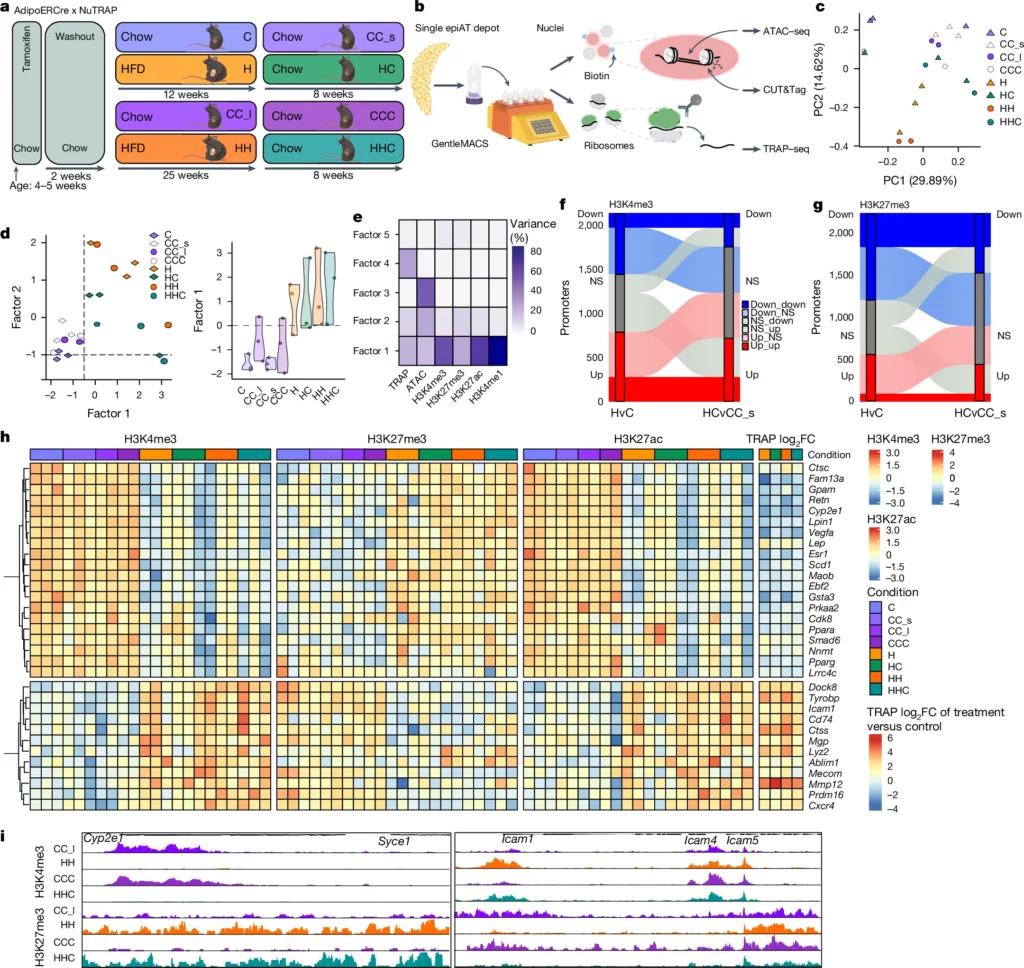
a, impostazione sperimentale dello studio WL in topi AdipoERCre x NuTRAP. b, flusso di lavoro di CUT&Tag, ATAC-seq e TRAP-seq accoppiati da un deposito di AT. I nuclei biotinilati e i ribosomi marcati con GFP sono stati isolati da tessuto congelato, tirati giù e sottoposti a CUT&Tag, ATAC-seq (nuclei) e TRAP-seq (ribosomi). c, PCA del translatomo (TRAP-seq) degli adipociti marcati di C, CC_s, CC_l, CCC, H, HH, HC e HHC. Ogni punto rappresenta un singolo replicato biologico. d, diagrammi MOFA che mostrano il raggruppamento dei campioni lungo i fattori latenti 1 e 2 (a sinistra) e la distribuzione dei valori del fattore 1 (a destra) negli adipociti etichettati. Ogni punto corrisponde a un replicato biologico. Per ogni replica, tutte le sei modalità sono rappresentate in un punto. e, Percentuale della varianza spiegata da ciascun fattore MOFA in una delle sei modalità. f, Dinamica dei promotori marcati in modo differenziato H3K4me3 (assey ) da H a HC. g, Dinamica dei promotori marcati in modo differenziato H3K27me3 (assey ) da H a HC. h, arricchimento scalare di H3K4me3 (a sinistra), H3K27me3 (al centro) e H3K27ac (a destra) in corrispondenza di promotori selezionati di geni e la FC log2di TRAP-seq da confronti con i controlli per gli stessi geni. i, distribuzione delle letture normalizzate di H3K4me3 e H3K27me3 ai loci Cyp2e1 e Icam1 nelle varie condizioni. La scala delle letture è stata eseguita per hPTM. NS, non significativo; v, contro.
Implicazioni cliniche e il fenomeno dello “yo-yo”
Il fenomeno dello “yo-yo”, ovvero il recupero di peso ciclico, è stato associato alla memoria epigenetica dell’obesità. Nei modelli murini, è stato osservato che gli animali precedentemente obesi guadagnano peso più rapidamente rispetto ai controlli quando esposti a diete ipercaloriche. Questo effetto è legato alla riattivazione di geni associati all’infiammazione e alla fibrosi nel tessuto adiposo, che compromettono la normale funzione metabolica.
Prospettive future: cancellare la memoria dell’obesità?
Le scoperte sulla memoria epigenetica aprono nuove strade terapeutiche. La possibilità di intervenire direttamente sui meccanismi epigenetici, ad esempio attraverso farmaci che modificano le istone o il DNA, potrebbe rappresentare un approccio rivoluzionario. Inoltre, strategie che combinano interventi nutrizionali, farmacologici e comportamentali potrebbero migliorare il mantenimento della perdita di peso, riducendo il rischio di ricadute.
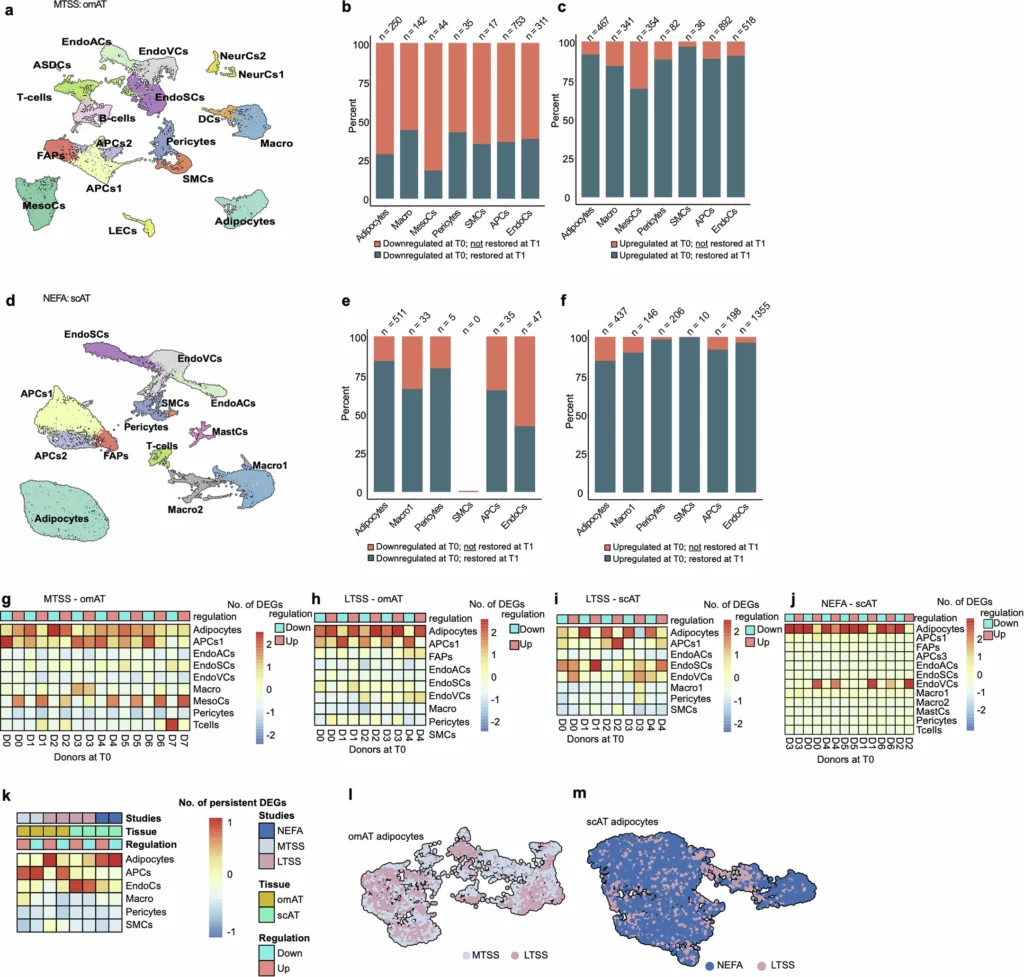
a, UMAP di 19.494 nuclei che rappresentano pool di omAT da soggetti magri (n= 5; 1 maschio, 4 femmine) e omAT accoppiati da T0 e T1 (n= 8 ciascuno; 2 maschi, 6 femmine) dallo studio MTSS. b,c Proporzione di cambiamenti trascrizionali conservati in tipi di cellule altamente abbondanti di omAT MTSS. d, UMAP di 31.721 nuclei che rappresentano pool di scAT di soggetti magri (n= 8; 8 femmine) e scAT accoppiati di T0 e T1 (n= 7 ciascuno; 7 femmine) dello studio NEFA. e,f Proporzione di cambiamenti trascrizionali conservati nei tipi di cellule altamente abbondanti di NEFA scAT. g-j Numero di DEG upregolati e downregolati per tipo di cellula del donatore obeso in scala per colonna a T0 per omAT (a sinistra) e scAT (a destra) dagli studi MTSS, LTSS e NEFA. k, Numero di geni persistentemente deregolati da T0 a T1 per tipo di cellula nei pool AT di tutti gli studi. l, UMAP di 4.958 nuclei che rappresentano gli adipociti di MTSS omAT e LTSS omAT (totale magro n= 10; totale T0/T1 n= 13). m, UMAP di 13.231 nuclei che rappresentano gli adipociti di NEFA scAT e LTSS scAT (totale magro n= 13; totale T0/T1 n= 12). Per l'identificazione dei DEG in b, c, e-k è stato utilizzato il Wilcoxon Rank Sum test, con valore di p aggiustato < 0,01 con il metodo di correzione di Bonferroni e FC > ±0,5. APCs, cellule progenitrici adipocitarie; ASDCs, cellule dendritiche AXL+; DCs, cellule dendritiche; EndoCs, cellule endoteliali; EndoACs, EndoCs arteriolari; EndoSCs, EndoCs peduncolari; EndoVCs, EndoCs venulari; LECs, cellule endoteliali linfatiche; FAPs, progenitori fibro-adipogenici; Macro, macrofagi; MastCs, mastociti; MesoCs, cellule mesoteliali; NeurCs, cellule simil-neuronali; SMCs, cellule muscolari lisce (vascolari).
In conclusione
La memoria epigenetica dell’obesità rappresenta un’importante sfida per il trattamento efficace e duraturo di questa condizione. Comprendere meglio i meccanismi sottostanti potrebbe trasformare l’approccio alla gestione del peso, offrendo nuove speranze per milioni di persone. La ricerca in questo campo è solo agli inizi, ma promette di aprire una nuova era nella lotta contro l’obesità e le sue complicanze.





