
“Il cancro e l’AIDS sono bestie educate: mangiano una ben misera razione di carne rispetto a quella che hai mangiato tu e non ti lasciano solo come tu tenti di fare con loro.”
— Fabrizio De André
La disregolazione della regolazione post-trascrizionale nell’espressione genica è un fenomeno complesso che può avere un impatto significativo sullo sviluppo del carcinoma mammario luminale B. In questo articolo, esploreremo i meccanismi di questa disregolazione e il suo ruolo nella progressione della malattia. Analizzeremo l’espressione differenziale di miRNA e mRNA, identificheremo le coppie miRNA-gene target coinvolte e discuteremo i risultati funzionali e i percorsi critici associati. Infine, esamineremo le implicazioni cliniche di questi risultati e discuteremo i futuri sviluppi nel campo della terapia mirata per il carcinoma mammario luminale B.
Introduzione alla disregolazione dell’espressione genica
L’introduzione alla disregolazione dell’espressione genica è un argomento fondamentale per comprendere i meccanismi che influenzano la regolazione post-trascrizionale nell’espressione genica e il suo coinvolgimento nel carcinoma mammario luminale B. La disregolazione dell’espressione genica si riferisce a alterazioni nei processi cellulari che controllano la trascrizione e la traduzione dei geni. Questa disregolazione può portare a cambiamenti nella quantità e nella qualità delle proteine prodotte, influenzando così il funzionamento normale delle cellule. Nel contesto del carcinoma mammario luminale B, la disregolazione dell’espressione genica è stata ampiamente studiata per identificare i miRNA e i geni target coinvolti nella progressione della malattia. L’analisi dell’espressione differenziale di miRNA e mRNA è uno strumento essenziale per identificare le alterazioni molecolari associate al carcinoma mammario luminale B e per comprendere meglio i meccanismi sottostanti alla sua patogenesi. La comprensione di questi meccanismi potrebbe aprire nuove prospettive per lo sviluppo di terapie mirate e personalizzate per questa forma di cancro al seno.
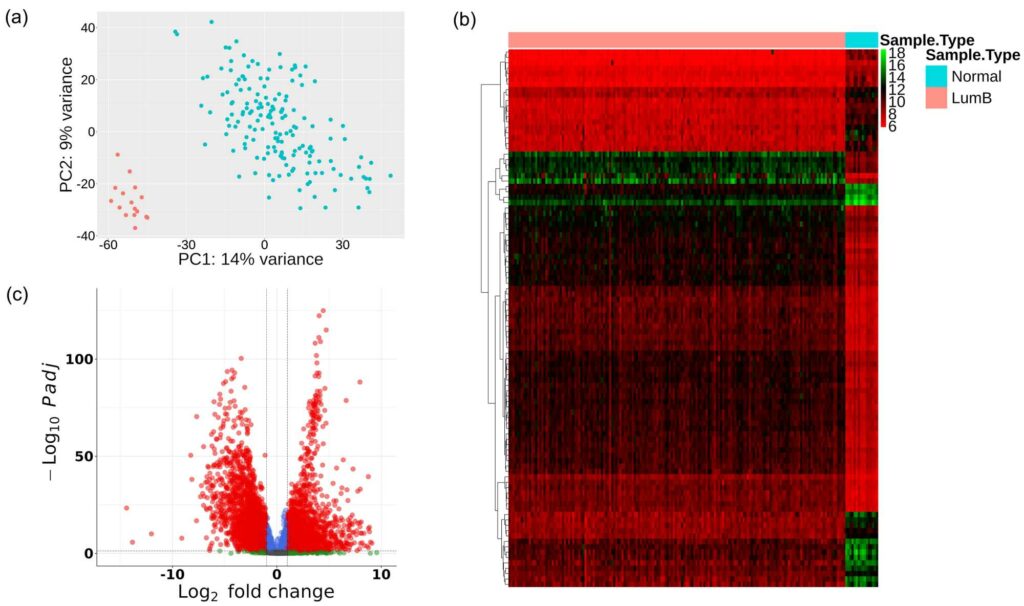
(a) Analisi delle componenti principali utilizzando i dati di espressione dell'mRNA. I punti blu rappresentano campioni di tumore luminale B; i punti rossi rappresentano campioni normali appaiati; PC1 rappresenta la componente principale 1; PC2 rappresenta la componente principale 2. (b) Heatmap dei primi 100 geni differenzialmente espressi. LumB rappresenta il sottotipo Luminal B. (c) Grafico a vulcano degli mRNA differenzialmente espressi. L'asse X rappresenta il log2 (fold change); l'asse Y rappresenta il -log10 (FDR); i punti rossi rappresentano i geni con espressione significativamente differenziale.
Analisi dell’espressione differenziale di miRNA e mRNA
L’analisi dell’espressione differenziale di miRNA e mRNA è un approccio fondamentale per comprendere la complessità della regolazione post-trascrizionale nell’espressione genica. Questo tipo di analisi consente di identificare i microRNA (miRNA) e i corrispondenti mRNA che mostrano variazioni significative nell’espressione tra campioni biologici diversi. Attraverso l’identificazione di queste differenze, è possibile ottenere una panoramica dettagliata delle vie di regolazione coinvolte nel carcinoma mammario luminale B. L’analisi dell’espressione differenziale dei miRNA può rivelare potenziali miRNA coinvolti nella patogenesi del carcinoma mammario luminale B, mentre l’analisi dell’espressione differenziale dei mRNA può fornire informazioni sulle alterazioni dei geni target. Questa analisi combinata aiuta a identificare le coppie miRNA-gene target che potrebbero svolgere un ruolo critico nella progressione del tumore. Inoltre, l’analisi dell’espressione differenziale di miRNA e mRNA può rivelare percorsi critici associati al carcinoma mammario luminale B, aprendo nuove possibilità per lo sviluppo di terapie mirate e prognosi personalizzate.
Identificazione delle coppie miRNA-Gene Target
L’identificazione delle coppie miRNA-Gene target è un processo cruciale per comprendere la regolazione post-trascrizionale dell’espressione genica nel carcinoma mammario luminale B. Questa analisi mira a identificare i geni bersaglio specifici che sono regolati dai miRNA e che potrebbero svolgere un ruolo chiave nello sviluppo e nella progressione del tumore. Attraverso l’utilizzo di approcci bioinformatici avanzati, è possibile predire le interazioni tra i miRNA e i loro geni bersaglio. Queste coppie miRNA-Gene target possono fornire informazioni preziose sulla funzione biologica dei miRNA coinvolti nel carcinoma mammario luminale B e suggerire nuovi percorsi critici associati alla malattia. L’identificazione accurata delle coppie miRNA-Gene target potrebbe inoltre aprire la strada a nuove terapie mirate basate sulla modulazione dei livelli di espressione dei miRNA o dei loro geni bersaglio. Tuttavia, ulteriori studi funzionali e validazioni sperimentali sono necessari per confermare il ruolo di queste coppie nel contesto clinico del carcinoma mammario luminale B.
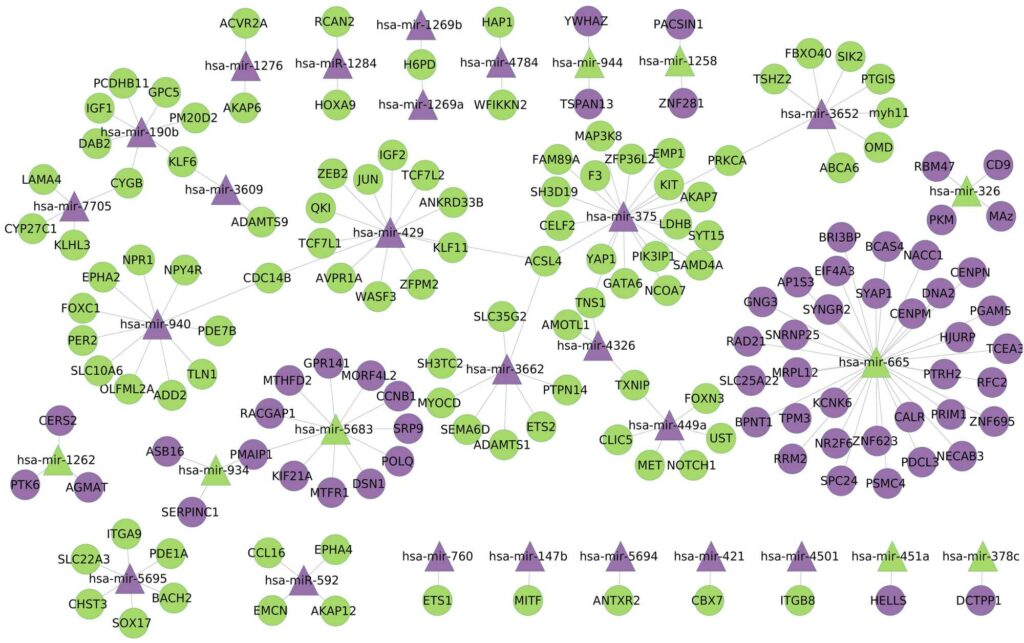
I nodi cerchiati rappresentano gli mRNA. I nodi triangolari rappresentano i miRNA; il colore viola rappresenta la sovraespressione e il colore verde la bassa espressione; i bordi rappresentano la correlazione tra l'espressione del miRNA e dell'mRNA.
Risultati funzionali e percorsi critici associati
I risultati funzionali e i percorsi critici associati alla disregolazione della regolazione post-trascrizionale nell’espressione genica nel carcinoma mammario luminale B rappresentano un importante punto di analisi nello studio di questa malattia. Gli studi condotti hanno evidenziato che l’alterata espressione di specifici miRNA e mRNA è coinvolta nella progressione e nell’aggressività del tumore mammario luminale B. In particolare, sono stati identificati diversi miRNA che agiscono come modulatori chiave della proliferazione cellulare, dell’invasione e della metastasi nel carcinoma mammario luminale B. Inoltre, è emerso che questi miRNA interagiscono con geni bersaglio coinvolti in importanti percorsi biologici, come la via di segnalazione del fattore di crescita epidermico (EGF) e la via di segnalazione del recettore degli estrogeni (ER). Questi risultati funzionali indicano una potenziale rilevanza clinica per lo sviluppo di nuove terapie mirate nel trattamento del carcinoma mammario luminale B. Tuttavia, ulteriori ricerche sono necessarie per comprendere appieno il ruolo di questi percorsi critici nella patogenesi del tumore mammario luminale B e per tradurre tali conoscenze in terapie efficaci per i pazienti affetti da questa malattia.
Discussione sulle implicazioni cliniche e futuri sviluppi
La discussione sulle implicazioni cliniche e futuri sviluppi della disregolazione della regolazione post-trascrizionale nell’espressione genica nel carcinoma mammario luminale B è di fondamentale importanza per comprendere l’impatto di questa disfunzione sulla malattia e per identificare potenziali obiettivi terapeutici. Le implicazioni cliniche si concentrano sulla possibilità di utilizzare la disregolazione dell’espressione genica come biomarcatore predittivo della risposta al trattamento e come possibile bersaglio terapeutico. Inoltre, le informazioni ottenute da questa ricerca possono contribuire allo sviluppo di nuovi farmaci mirati che agiscano sulle vie di regolazione post-trascrizionale. Per quanto riguarda i futuri sviluppi, è necessario approfondire ulteriormente la comprensione dei meccanismi molecolari coinvolti nella disregolazione dell’espressione genica e studiare in dettaglio gli effetti delle varie coppie miRNA-gene target coinvolte nel carcinoma mammario luminale B. Questi studi potrebbero portare a nuove scoperte che potrebbero migliorare la diagnosi, la prognosi e il trattamento di questa forma di cancro al seno.
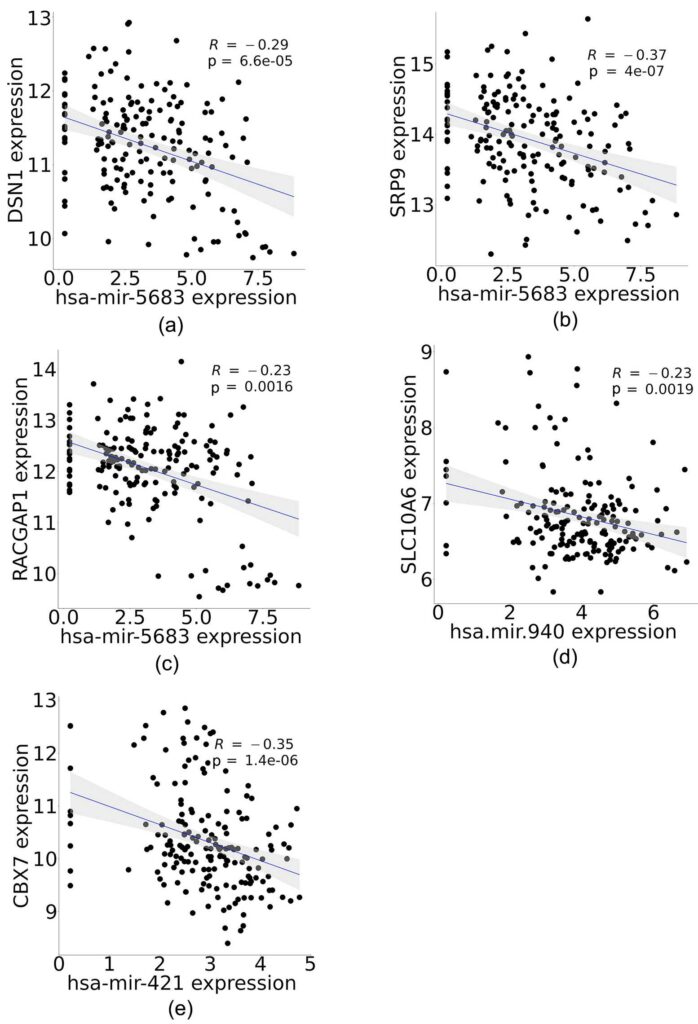
Grafici di dispersione che mostrano la correlazione tra i geni target prognostici ((a)-(d)) e i miRNA che li bersagliano, (a) DSN1, (b) SRP9, (c) RACGAP1 e (d) SLC10A6. (e) Grafico di dispersione che mostra la correlazione tra il microRNA prognostico hsa-mir-421 e il suo gene bersaglio CBX7. L'asse X rappresenta i valori di espressione normalizzati del miRNA; l'asse Y rappresenta i valori di espressione normalizzati dell'mRNA. R è il coefficiente di correlazione di Spearman.
Ulaganathan, K. , Alipeddi, R. and Hanumanth, S. (2024) Identifying microRNA-Target Gene Pairs in Luminal B Breast Cancer Using Integrated Analysis of miRNA and Transcriptome Profiles. Advances in Breast Cancer Research, 13, 69-100. doi: 10.4236/abcr.2024.134008.
In conclusione…
In definitiva, lo studio sulla disregolazione della regolazione post-trascrizionale nell’espressione genica nel carcinoma mammario luminale B ha fornito importanti risultati che contribuiscono alla comprensione dei meccanismi molecolari coinvolti nello sviluppo e nella progressione di questa forma di cancro al seno. L’analisi dell’espressione differenziale di miRNA e mRNA ha permesso di identificare le coppie miRNA-gene target coinvolte nel processo patologico. I risultati funzionali e i percorsi critici associati hanno svelato nuove informazioni sulla biologia del carcinoma mammario luminale B. Tuttavia, rimane ancora molto da scoprire e approfondire in questo campo. Un punto aperto per la riflessione riguarda le implicazioni cliniche di queste scoperte e come possano essere tradotte in nuove strategie terapeutiche mirate. Inoltre, futuri sviluppi potrebbero includere l’indagine di ulteriori fattori regolatori coinvolti nell’espressione genica, così come l’utilizzo di tecnologie avanzate per comprendere meglio la complessità dei meccanismi molecolari sottostanti al carcinoma mammario luminale B.





